Se trata de péptidos antimicrobianos que podrían eliminar bacterias resistentes e impulsar nuevos medicamentos para tratar infecciones. El estudio fue publicado en la revista Cell Host & Microbe. Los detalles
La idea de reintroducir organismos extintos en ambientes existentes ha capturado la imaginación pública y científica, planteando profundas cuestiones éticas y ecológicas. Ahora, una nueva investigación presenta la eliminación de la extinción molecular como un marco de descubrimiento de antibióticos. La de-extinción molecular es la resurrección de moléculas de vida extintas: ácidos nucleicos, proteínas y otros compuestos que ya no están codificados por organismos vivos.
Si bien el beneficio social de la extinción de organismos aún se desconoce y es polémico, los desafíos técnicos, como la cobertura genómica incompleta, siguen siendo importantes. Un equipo de bioingenieros de la Universidad de Pensilvania ha utilizado la inteligencia artificial (IA) para resucitar moléculas.
Para realizar esta desextinción, aplicaron métodos computacionales sobre proteínas tanto de humanos modernos (Homo sapiens) como de neandertales (Homo neanderthalensis) y los denisovanos (población de humanos primitivos que vivieron en Asia y estuvieron relacionados a los neandertales de forma lejana).
Esto le permitió a los autores identificar moléculas que pueden matar bacterias que causan enfermedades, un hallazgo que podría inspirar nuevos medicamentos para tratar infecciones humanas. “Nos motiva la idea de recuperar moléculas del pasado para abordar los problemas que tenemos hoy”, explicó César de la Fuente, coautor del estudio y bioingeniero de la Universidad de Pensilvania en Filadelfia. El trabajo se publicó en Cell Host & Microbe.
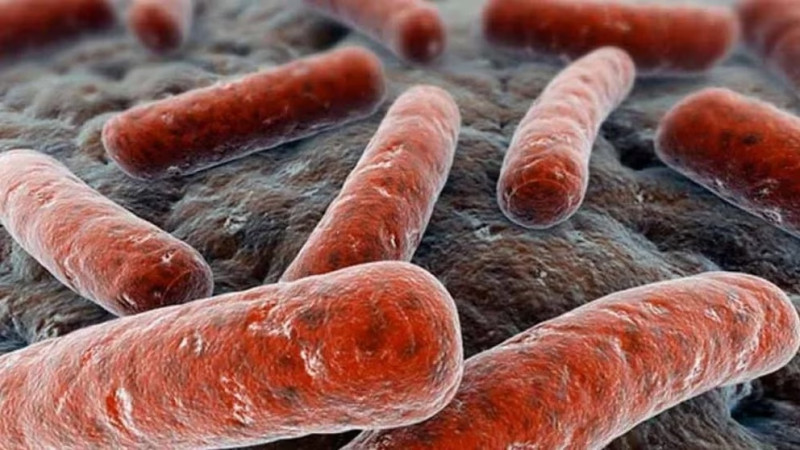
El desarrollo de antibióticos se ha ralentizado en las últimas décadas y la mayoría de los prescritos en la actualidad han estado en el mercado durante más de 30 años. Mientras tanto, las bacterias resistentes a los antibióticos van en aumento, por lo que pronto se necesitará una nueva ola de tratamientos. Muchos organismos producen subunidades proteicas cortas llamadas péptidos que tienen propiedades antimicrobianas. Un puñado de péptidos antimicrobianos, la mayoría de los cuales fueron aislados de bacterias, ya están en uso clínico.
Un nuevo camino
Las proteínas de especies extintas podrían ser un recurso sin explotar para el desarrollo de antibióticos, una realización a la que de la Fuente y sus colaboradores llegaron gracias, en parte, al cine. “Empezamos a pensar realmente en Jurassic Park -relató-. En lugar de devolver la vida a los dinosaurios, como hicieron los científicos en la película, al equipo se le ocurrió una idea más factible: ¿Por qué no traer de vuelta las moléculas?”.
Para ello los especialistas entrenaron un algoritmo de IA para reconocer sitios en proteínas humanas donde se sabe que se cortan en péptidos. Para encontrar nuevos péptidos, el equipo aplicó su algoritmo a secuencias de proteínas disponibles públicamente (mapas de los aminoácidos en una proteína) de H. sapiens, H. neanderthalensis y Denisovans.
Luego, utilizaron las propiedades de los péptidos antimicrobianos descritos anteriormente para predecir cuál de los nuevos podría matar bacterias. “Encontrar y probar candidatos a fármacos utilizando IA lleva unas semanas. Por el contrario, se necesitan de tres a seis años usando métodos más antiguos para descubrir un solo antibiótico nuevo”, explica De la Fuente.
Probaron docenas de péptidos para ver si podían matar bacterias en placas de laboratorio. Luego seleccionaron seis potentes, cuatro de H. sapiens, uno de H. neanderthalensis y uno de Denisovans, y se los administraron a ratones infectados con la bacteria Acinetobacter baumannii, una causa común de infecciones hospitalarias en humanos. Los seis péptidos detuvieron el crecimiento en el músculo del muslo, pero ninguno mató a la bacteria. Cinco de las moléculas mataron las bacterias que crecían en los abscesos de la piel, pero recibieron un fuerte golpe. Aún así, las dosis utilizadas fueron extremadamente altas.
“Ajustar las moléculas más exitosas podría crear versiones más efectivas -señaló De la Fuente-. Asimismo, alterar el algoritmo podría mejorar la identificación de péptidos antimicrobianos, con menos falsos positivos. Aunque el algoritmo que usamos no produjo moléculas sorprendentes, creo que el concepto y el marco representan una vía completamente nueva para pensar sobre el descubrimiento de fármacos”. Completaron el equipo de investigación Jacqueline Maasch, Marcelo Torres y Marcelo Melo.
Fuente: Infobae.







Dejanos tu comentario
Su comentario estará disponible a la brevedad.